使用正则表达式拆分列中的值
我有data.frame有两列,如下所示
dat
ID Details
id_1 box1_homodomain gn=box1 os=homo sapiens p=4 se=1
id_2 sox2_plurinet gn=plu os=mus musculus p=5 se=3
我想在所有ID的“详细信息”列中拆分“os = xxx”和gn =“yyy”,并将其打印如下:
Id Description gn os
Îd_1 box1_homodomain box1 homo sapiens
Id_2 sox2_plurinet plu mouse musculus
我尝试在R中使用gsub方法但是我无法将os = homo sapiens和gn = box1分成各自的列。我使用的以下R代码
dat$gn=gsub('^[gn=][A-z][A-z]`,dat$Details)
dat$os=gsub('^[os=][A-z][A-z]`,dat$Details)
任何人都可以告诉我哪些错误以及如何纠正错误。请帮助我。
提前致谢
3 个答案:
答案 0 :(得分:5)
这是tidyr的一个选项:
library(tidyr)
# specify the new column names:
vars <- c("Description", "gn", "os")
# then separate the "Details" column according to regex and drop extra columns:
separate(dat, Details, into = vars, sep = "[A-Za-z]+=", extra = "drop")
# ID Description gn os
#1 id_1 box1_homodomain box1 homo sapiens
#2 id_2 sox2_plurinet plu mus musculus
答案 1 :(得分:4)
1)sub和gsub 要使用sub和gsub执行此操作,请尝试此操作。请注意,每个正则表达式应与dat$Details的所有正则表达式匹配,以便在我们将其替换为捕获组时,仅保留捕获组。对于问题评论中的dat$GO,我们会移除所有内容,但不包括P:,请用逗号替换所有;P,并删除P:之后删除分号和所有内容。同样适用于F和C:
data.frame(dat[1],
Description = sub(" .*", "", dat$Details),
gn = sub(".*gn=(.*) os=.*", "\\1", dat$Details),
os = sub(".*os=(.*) p=.*", "\\1", dat$Details),
P = gsub("P:|;.*", "", gsub(";P:", ",", sub(".*?P:", "P:", dat$GO))),
F = gsub("F:|;.*", "", gsub(";F:", ",", sub(".*?F:", "F:", dat$GO))),
C = gsub("C:|;.*", "", gsub(";C:", ",", sub(".*?C:", "C:", dat$GO))))
,并提供:
ID Description gn os P F C
1 id_1 box1_homodomain box1 homo sapiens p_1,p_2 F_1 C_1,C_2
2 id_2 sox2_plurinet plu mus musculus p_1 F_1,F_2 C_1
2)read.pattern dat$Details的{{1}} (link)处理read.pattern稍微容易一点,因为可以定义< em>单个正则表达式,其捕获组代表感兴趣的字段。通过使用dat$GO gsubfn package提取P:...字段,然后将其与strapplyc连接在一起,可以简化paste的处理(与{{1}相似}}和F字段):
C,并提供:
library(gsubfn)
Sub <- function(string, pat) sapply(strapplyc(string, pat), paste, collapse = ",")
DF <- read.pattern(text = as.character(dat$Details),
pattern = "(.*) gn=(.*) os=(.*) p=",
col.names = c("Description", "gn", "os"),
as.is = TRUE)
cbind(dat[1], DF,
P = Sub(dat$GO, "P:(.*?);"),
F = Sub(dat$GO, "F:(.*?);"),
C = Sub(dat$GO, "C:(.*?);"))
以下是 ID Description gn os P F C
1 id_1 box1_homodomain box1 homo sapiens p_1,p_2 F_1 C_1,C_2
2 id_2 sox2_plurinet plu mus musculus p_1 F_1,F_2 C_1
中使用的正则表达式的可视化:
read.pattern 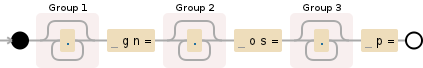
备注
1)如果(.*) gn=(.*) os=(.*) p=
列已经是字符,我们可以省略dat$Details。如果结果中包含as.character列,我们也可以省略as.is=TRUE。
2)问题中的示例输出为factor,但输入为mouse。我们假设在两种情况下都应该是mus。
3)我们将此用于mus:
dat将来请在问题中发布dat <-
structure(list(ID = c("id_1", "id_2"),
Details = c("box1_homodomain gn=box1 os=homo sapiens p=4 se=1",
"sox2_plurinet gn=plu os=mus musculus p=5 se=3"),
GO = c("P:p_1;P:p_2;F:F_1;C:C_1;C:C_2; ",
"P:p_1;F:F_1;F:F_2;C:C_1;")), .Names = c("ID", "Details",
"GO"), class = "data.frame", row.names = c(NA, -2L))
的结果。
答案 2 :(得分:0)
您也可以使用正则表达式捕获组。
每个捕获组匹配可以用例如提取。 stri_match_first_regex包中的stringi函数。
dat <- data.frame(
ID=c("id_1", "id_2"),
details=c("box1_homodomain gn=box1 os=homo sapiens p=4 se=1", "sox2_plurinet gn=plu os=mus musculus p=5 se=3")
)
library(stringi)
res <- stri_match_first_regex(dat$details, "^(.+) gn=(.+) os=(.+) p=.*$")
res[,1] <- dat$ID
res <- as.data.frame(res)
names(res) <- c("ID", "Description", "gn", "os")
res
## ID Description gn os
## 1 1 box1_homodomain box1 homo sapiens
## 2 2 sox2_plurinet plu mus musculus
- 我写了这段代码,但我无法理解我的错误
- 我无法从一个代码实例的列表中删除 None 值,但我可以在另一个实例中。为什么它适用于一个细分市场而不适用于另一个细分市场?
- 是否有可能使 loadstring 不可能等于打印?卢阿
- java中的random.expovariate()
- Appscript 通过会议在 Google 日历中发送电子邮件和创建活动
- 为什么我的 Onclick 箭头功能在 React 中不起作用?
- 在此代码中是否有使用“this”的替代方法?
- 在 SQL Server 和 PostgreSQL 上查询,我如何从第一个表获得第二个表的可视化
- 每千个数字得到
- 更新了城市边界 KML 文件的来源?