识别&从PCA&中删除异常值QQ情节
我有一个132 x 107的数据集,包括2个患者类型 - (患者1的33个)和(患者2的99个)。
我正在寻找异常值,所以我在数据集上运行了pca并使用以下命令完成了前4个组件的qqplots
pca = prcomp(data, scale. = TRUE)
plot(pca$x, pch = 20, col = c(rep("red", 33), rep("blue", 99)))
当我使用:
执行第二个组件的qqplot时qqPlot(pca$x[,2],pch = 20, col = c(rep("red", 33), rep("blue", 99)))
下图显示了2个明确的异常值 - 左下角的红点是患者1。
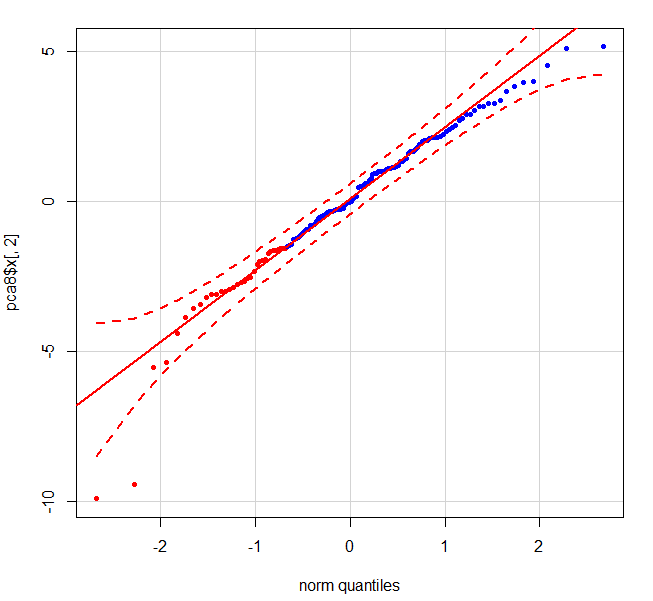
有没有直接的方法来计算数据中这些点的索引,以便将它们删除?
3 个答案:
答案 0 :(得分:6)
出于某种原因,我不相信支持识别方法
在car包中(qqPlot()的来源)
让我们来看看USArrests数据的PCA ......
pca <- prcomp(USArrests)
使用qqPlot的情节很容易。
require(car)
qqPlot(pca$x[,2],pch = 20, col = c(rep("red", 33), rep("blue", 99)))
但是,qqPlot()不允许通过identify()进行点选择。
identify(qqPlot(pca$x[,2],pch = 20, col = c(rep("red", 33), rep("blue", 99))))
# numeric(0)
但是,您可以在qqnorm()包中使用stats。
identify(qqnorm(pca$x[,2],pch = 20, col = c(rep("red", 33), rep("blue", 99))))
这将产生一个不太复杂的图形,但您应该能够通过qqline()(也在stats)手动添加线和置信区间以及更多的数学运算。
答案 1 :(得分:3)
您可以在R中尝试identify方法。通常,运行
identify(qqPlot(pca$x[,2],pch = 20, col = c(rep("red", 33), rep("blue", 99))))
并左键单击要识别的点。分数向量中的点索引应与原始数据中的相同。
答案 2 :(得分:0)
您还可以使用fviz_pca_ind()库中的factoextra函数来可视化影响,如下所示:
require(factoextra)
pca = prcomp(mydata)
fviz_pca_ind(pca,
col.ind = "contrib", # Color by contribution
gradient.cols = c("#00AFBB", "#E7B800", "#FC4E07") #assign gradient
)
这会自动标记个人,并根据他们的影响为他们着色。
相关问题
最新问题
- 我写了这段代码,但我无法理解我的错误
- 我无法从一个代码实例的列表中删除 None 值,但我可以在另一个实例中。为什么它适用于一个细分市场而不适用于另一个细分市场?
- 是否有可能使 loadstring 不可能等于打印?卢阿
- java中的random.expovariate()
- Appscript 通过会议在 Google 日历中发送电子邮件和创建活动
- 为什么我的 Onclick 箭头功能在 React 中不起作用?
- 在此代码中是否有使用“this”的替代方法?
- 在 SQL Server 和 PostgreSQL 上查询,我如何从第一个表获得第二个表的可视化
- 每千个数字得到
- 更新了城市边界 KML 文件的来源?