如何在R中删除具有特定模式格式数据框的字符
我试图删除所有以模式开头的字符" Gm"从我的data.frame的最后一栏
我的data.frame看起来像这样
level logp chr start end CNA Genes
3 1.4 3 100 110 gain Gm5852,Gm5773,Tdpoz4,Tdpoz3,Gm911
4 18.10 3 962 966 gain Fcgr1,Terc,Gm5703
结果看起来应该是这样的
level logp chr start end CNA Genes
3 1.4 3 100 110 gain Tdpoz4,Tdpoz3
4 18.10 3 962 966 gain Fcgr1,Terc
2 个答案:
答案 0 :(得分:5)
这使用单个gsub删除不需要的部分:
Genes <- c("Gm5852,Gm5773,Tdpoz4,Tdpoz3,Gm911", "Fcgr1,Terc,Gm5703") # test data
gsub(",?Gm[^,]*,?", "", Genes)
,并提供:
[1] "Tdpoz4,Tdpoz3" "Fcgr1,Terc"
以下是正则表达式的可视化:
,?Gm[^,]*,?
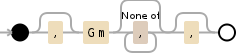
答案 1 :(得分:2)
给定数据框d:
d$Genes_new <- sapply(strsplit(as.character(d$Genes),split=','),function(s) paste(s[!grepl('^Gm',s)],collapse=','))
# level logp chr start end CNA Genes Genes_new
#1 3 1.4 3 100 110 gain Gm5852,Gm5773,Tdpoz4,Tdpoz3,Gm911 Tdpoz4,Tdpoz3
#2 4 18.1 3 962 966 gain Fcgr1,Terc,Gm5703 Fcgr1,Terc
此处,strsplit(as.character(d$Genes),split=',')为每行创建逗号分隔的基因名称列表,sapply适用于此列表的每个元素,该函数排除从Gm开始的所有基因名称({{1}并且连接剩余的基因(s[!grepl('^Gm',s)]。
相关问题
最新问题
- 我写了这段代码,但我无法理解我的错误
- 我无法从一个代码实例的列表中删除 None 值,但我可以在另一个实例中。为什么它适用于一个细分市场而不适用于另一个细分市场?
- 是否有可能使 loadstring 不可能等于打印?卢阿
- java中的random.expovariate()
- Appscript 通过会议在 Google 日历中发送电子邮件和创建活动
- 为什么我的 Onclick 箭头功能在 React 中不起作用?
- 在此代码中是否有使用“this”的替代方法?
- 在 SQL Server 和 PostgreSQL 上查询,我如何从第一个表获得第二个表的可视化
- 每千个数字得到
- 更新了城市边界 KML 文件的来源?